1. 本公司专业技术团队拥有多年的walking技术经验,尤其在TAIL-PCR(交错式热不对称PCR)长期的实验中积累了丰富经验; 2. 高效快速地最大可能保证您获得想要的序列。 3. 针对含有高级结构的未知序列,摸索出一套快速高效的独特方法。
请在线咨询或直接联系17720555749
技术简介
染色体步移(Chromosome walking),又称为基因组步移(Genome walking),是指由生物基因组或基因组文库中的已知序列出发,逐步探知其旁邻的未知序列或与已知序列呈线性关系的目标序列的方法。对于已经完成基因组测序的模式生物种(如人、小鼠、线虫、水稻、拟南芥等),可直接从数据库中轻松的找到已知序列的侧翼序列。但是目前为止,自然界中大多数生物基因组DNA序列仍未知,要想知道一个已知区域两侧的DNA序列,染色体步移无疑是一种非常有效的方法。
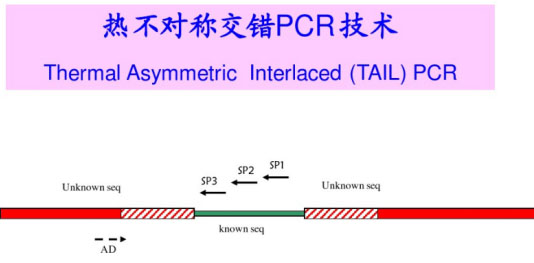
TAIL-PCR (Thermal Asymmetric Interlaced PCR),即热不对称PCR,又叫巢式PCR,克服了传统染色体步移法(如构建文库步移法、反向PCR法、连接接头法等)操作复杂、非特异性扩增、连接效率低等弊端。其原理是,根据已知 DNA 序列,分别设计三条同向且退火温度较高的特异性引物(SP Primer),与经过独特设计的退火温度较低的简并引物进行热不对称 PCR 反应。通常情况下,其中至少有一种兼并引物可以与特异性引物之间利用退火温度的差异进行热不对称 PCR 反应,一般通过三次巢式 PCR 反应即可获取已知序列的侧翼序列。如果一次实验获取的长度不能满足实验要求时,还可以根据第一次步移获取的序列信息,继续进行侧翼序列获取。
应用领域
1. 鉴定T-DNA或转座子的插入位点,鉴定转基因技术所导致的外源基因的插入位点; (2)根据基因的已知片段、EST或插入的转座子序列克隆目的基因,分离基因的启动子及调控元件;
2. 用于人工染色体PAC、YAC和BAC的片段搭接;
3. 构建图位克隆中的重叠群;
4. 转化标记辅助育种中的STS或SCAR标记等
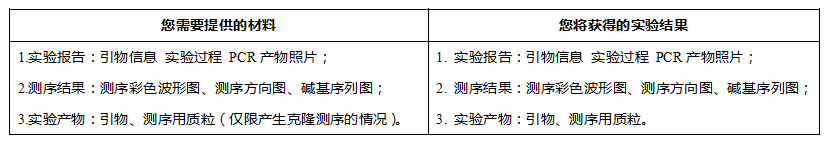
服务内容

服务流程
1.已知序列的验证
根据客户提供的已知序列设计引物,以材料的基因组DNA为模板进行PCR扩增,以验证已知序列是否在模板中存在。
2. 5' 或3' 未知序列的获取
根据已知序列设计3条高退火温度的特异性下游或上游引物,同时使用低退火温度的简并引物,利用特异性引物与简并引物之间退火温度的差异进行不对称PCR,通过三次巢式PCR获取侧翼序列。
3. 对获取的序列进行验证
获取侧翼未知序列后,我们将分别在已知和未知序列上设计引物进行PCR扩增并对PCR产物进行测序,以验证获取序列的真实性。

样品与实验结果
实验结果示例